安捷伦SureSelect靶向序列捕获系统
产品名称: 安捷伦SureSelect靶向序列捕获系统
英文名称: Agilent SureSelect
产品编号:
产品价格: 询价
产品产地: null
品牌商标: null
更新时间: null
使用范围: null
安捷伦科技有限公司Agilent
- 联系人 :
- 地址 : 上海市虹口区四川北路1350号16楼
- 邮编 :
- 所在区域 : 上海
- 电话 : 点击查看
- 传真 : 点击查看
- 邮箱 : mary.li@agilent.com
安捷伦SureSelect靶向序列捕获系统
解决研究中的费用瓶颈问题,发挥高通量测序的全部力量
最新推出“人全外显子捕获”试剂盒!全面支持Solexa和SOLiD测序平台!
Nature Biotech封面文章:液相序列捕获技术与高通量测序
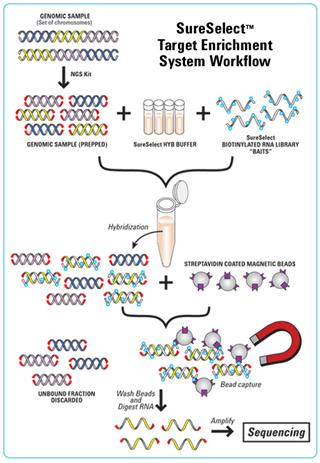
安捷伦的SureSelect靶向序列捕获系统可以显著降低下一代测序技术(next-generation sequencing)的成本,并且可以极大程度的提高测序效率。SureSelect是一套基于安捷伦卓越的寡核苷酸合成技术的液相靶向序列捕获系统,是一个适用于任何规模测序研究项目的可缩放平台。完全不同于以往其它的序列捕获方法,SureSelect系统仅仅需要3 µg的基因组DNA,这意味着研究者即使对珍贵样本也可以进行高通量的平行靶向测序!
图 1. SureSelect 靶向序列捕获系统操作流程
借助eArray完成定制的序列捕获系统,可能包含完全不同的RNA “诱饵”寡核苷酸序列,它们的表现又会怎样?为了回答这个问题,安捷伦进行了大量的研究验证工作。针对同一样本的不同基因组序列区域,我们设计了多种不同的序列捕获方案,进行多组平行对照实验,利用测序结果中靶向序列读出的比率作为富集效率的评判指标。如图2所示,8种不同的定制序列捕获方案所获得的靶向序列读出的比率在40-80之间,意味着300-7400倍的富集效率,充分验证了SureSelect系统的优异表现。
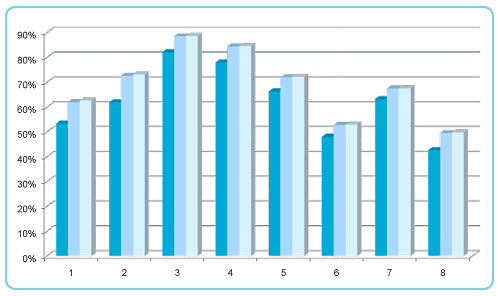 |
图2. SureSelect靶向序列捕获系统与定制设计策略
不管序列捕获设计方法的特异性如何优异,因为研究人员对基因组DNA进行随机剪切,所以对靶向序列相邻区域的捕获仍然不能完全避免。但是,如图2所示,安捷伦的SureSelect系统拥有极佳的序列捕获特异性,对离开靶向序列区域200 bp以外的序列几乎没有捕获。
|
衡量DNA测序技术的另外一个重要指标是测序数据的覆盖深度。较高的覆盖深度,往往也意味着更好的辨识序列多态性的能力。图3的测序结果表明,超过80的序列都可以获得20x以上的覆盖深度,因此完全可以轻松完成利用重新测序方法对基因组特定区域中SNP的辨识。
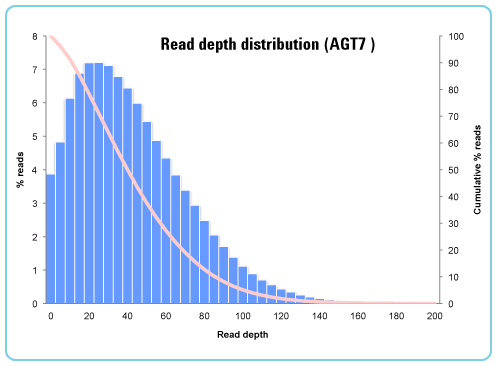 |
图 3. SureSelect 靶向序列捕获系统的覆盖深度
产品信息
|
|
SureSelect液相系统 |
SureSelect固相系统 |
|
研究 |
10-1,000样本 |
1-10 样本 |
|
DNA起始量 |
1-3 µg |
1-20 µg* |
|
可捕捉的序列长度 (定制试剂盒。不收设计费) |
6。6+ Mb |
1+ Mb |
最新推出“人全外显子捕获”试剂盒!全外显子序列(权威CCDS数据库)和>1,000非编码RNA,一次试验,一网打尽!
,